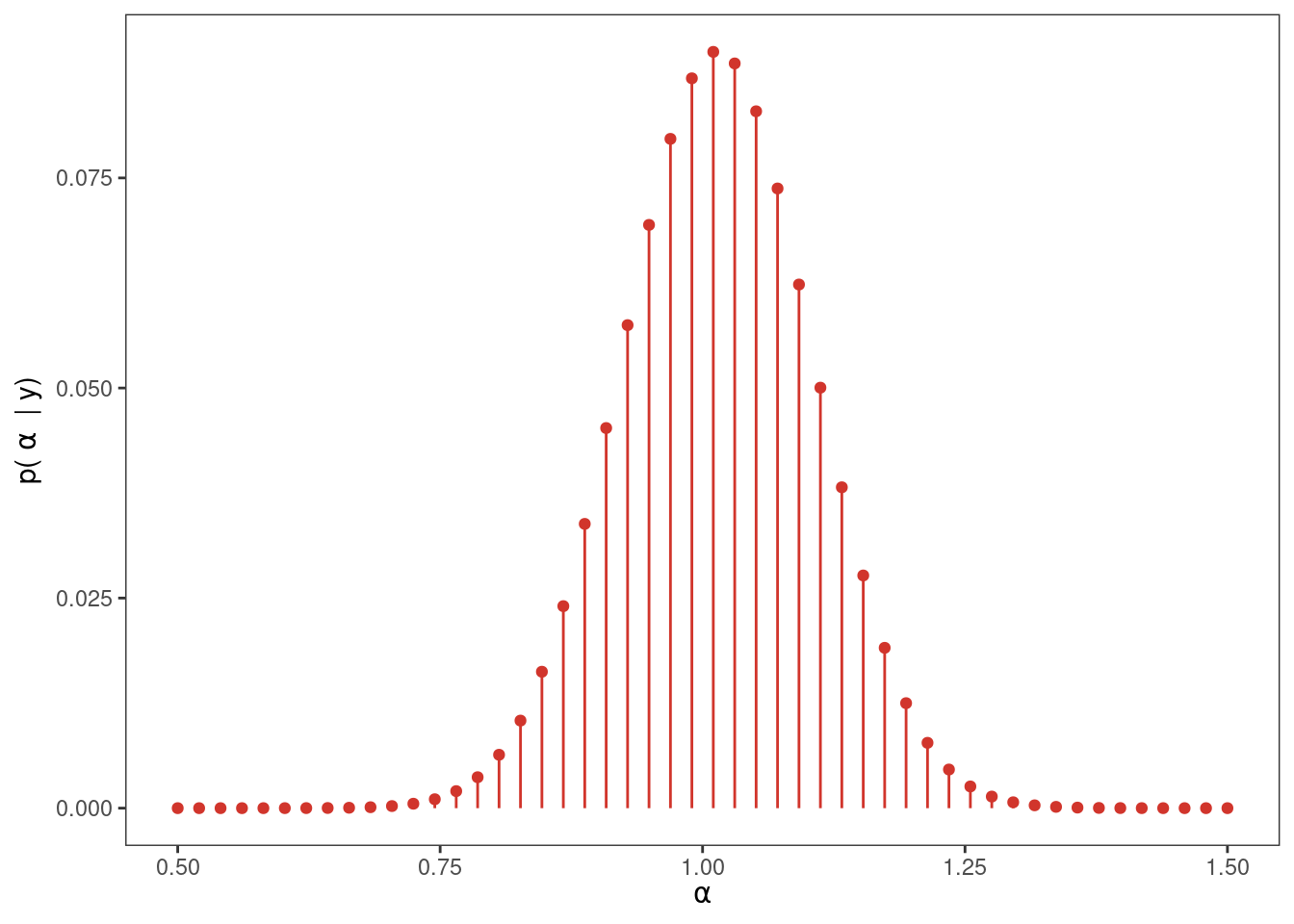
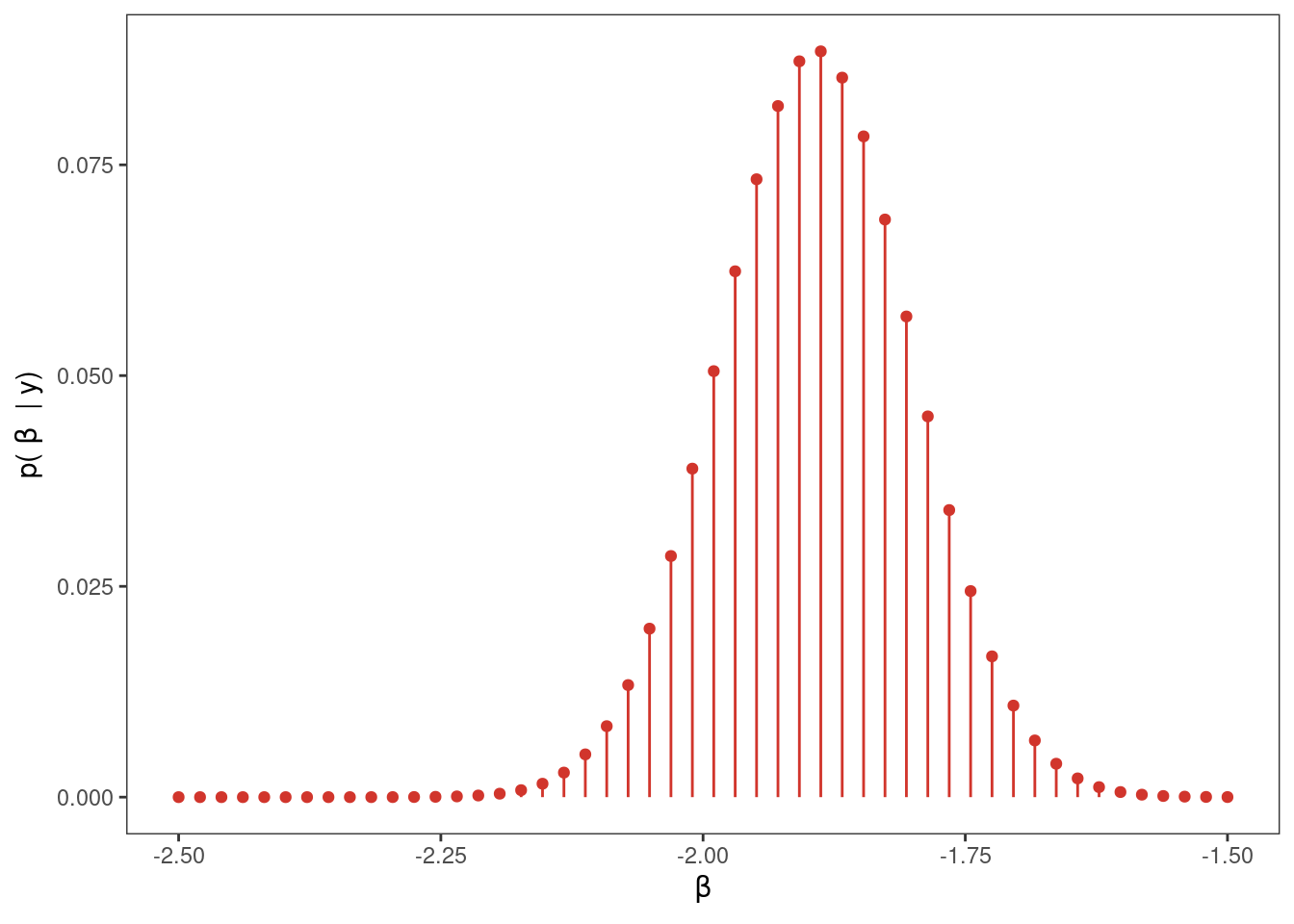
library(dplyr)
library(ggplot2)
set.seed(121195)
# Generación de datos
alpha <- 1
beta <- -2
sigma <- 0.8
n <- 80
x <- rnorm(n)
y <- rnorm(n, alpha + beta * x, sigma)
df <- data.frame(x = x, y = y)
# Crear grilla para alfa y beta
grid_a <- seq(0.5, 1.5, length.out = 50)
grid_b <- seq(-2.5, -1.5, length.out = 50)
# Crear todas las combinaciones entre los valores de las dos grillas
grid_df <- expand.grid(grid_a, grid_b)
# Utilizar nombres indicativos
names(grid_df) <- c("a", "b")
# Crear vectores que van a contener los valores de la
# función de verosimilitud y del posterior
likelihood <- numeric(nrow(grid_df))
posterior <- numeric(nrow(grid_df))
# Calcular la función de verosimilitud en cada punto
for (i in seq_along(likelihood)) {
likelihood[i] <- prod(dnorm(y, grid_df$a[i] + grid_df$b[i] * x, sigma))
}
# Calcular el posterior en cada punto
posterior <- (
likelihood
* dnorm(grid_df$a, mean = 0, sd = 1.5) # alpha ~ Normal(0, 1.5)
* dnorm(grid_df$b, mean = 0, sd = 2) # beta ~ Normal(0, 2)
)
# Escalar el posterior para que sea propio
posterior <- posterior / sum(posterior)
# Incorporar likelihood y posterior al data frame
grid_df$likelihood <- likelihood
grid_df$posterior <- posterior
# Graficar con ggplot
ggplot(grid_df, aes(x = a, y = b)) +
geom_raster(aes(fill = posterior)) +
stat_contour(aes(z = posterior), col = "white", bins = 5) +
geom_point(x = alpha, y = beta, color = "black", fill = "red", size = 3, pch = 21) +
labs(x = expression(alpha), y = expression(beta)) +
viridis::scale_fill_viridis() +
guides(fill = guide_colourbar(barheight = unit(4.2, "in"))) +
scale_x_continuous(expand = c(0, 0)) +
scale_y_continuous(expand = c(0, 0))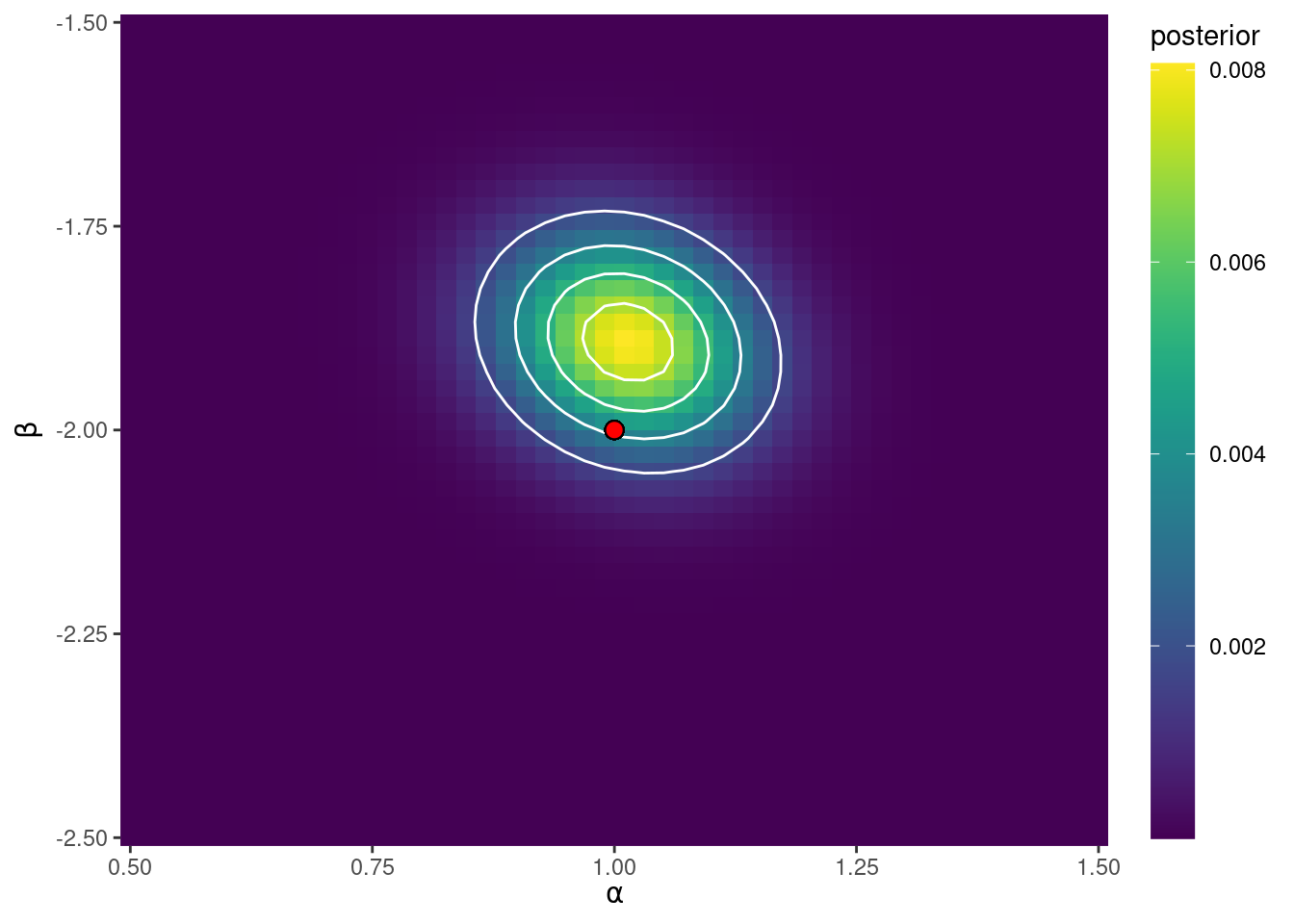
El punto indica los valores de \(\alpha\) y \(\beta\) utilizados para generar los datos.